(图文|杜泽臻 编辑|信息 审核|焦文标)近日,家庭教师av 果蔬园艺作物种质创新与利用全国重点实验室、湖北洪山实验室焦文标教授团队在Molecular Plant发表题为“Varigraph: an accurate and widely applicable pangenome graph-based variant genotyper for diploid and polyploid genomes”的研究论文。该研究开发了一种广泛适用于各类二倍体和多倍体基因组的遗传变异分型算法Varigraph。该算法基于最新图形泛基因组理论,利用短读长测序数据,即可实现各类遗传变异(SNP/Indel/SV)的精确分型。该研究为种质资源精准鉴定、基因组辅助育种等提供了精准、高效的算法框架和研究工具。

原文链接: //doi.org/10.1016/j.molp.2025.08.001
以基因组技术辅助的现代育种体系通常需要精准、快速鉴定种质资源的基因型信息。通过全基因组测序、比对测序数据到参考基因组,并利用基因组学算法鉴定变异位点,能完成目标个体的基因型鉴定。传统算法由于依赖单一线性参考基因组,导致部分测序数据比对错误或无法比对。基于泛基因组图谱的方法能解决单一参考序列带来的偏差,提升变异分型的效果。然而,现有方法多针对人类基因组开发,在处理大型或重复序列较高的植物泛基因组图谱时存在较大挑战,在多倍体基因组中表现得尤为明显。
针对上述不足,该研究开发了基于泛基因组图的变异分型新算法。Varigraph利用基因组序列和遗传变异集构建泛基因组图(Pangenome Graph)。通过比较泛基因组图中变异位点的Kmer和基因组测序读段的Kmer,确定变异位点的基因型。Varigraph算法的主要创新点在于:其一,同时考虑唯一性Kmer和低频重复性Kmer,以提升重复区域的变异分型效果;其二,利用计数布隆过滤器、位图存储、GPU加速等策略,降低内存消耗、提升运行速度;其三,构建多倍体变异分型模型,适用于从二倍体到八倍体的各种倍性基因组。
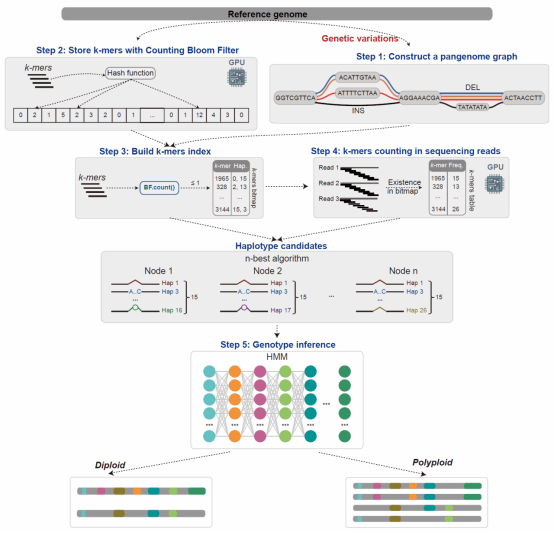
图1 Varigraph的算法流程
在二倍体基因组拟南芥、水稻、柚、玉米上的测试表明,Varigraph在各类变异分型上的效果均优于其他软件。Varigraph在重复区域的变异分型效果显著好于其他方法;相比于非重复区域,Varigraph的变异分型F值略有下降,而其他软件则下降较为明显。另外,Varigraph在结构变异的分型F-值明显高于其他方法。综上,表明Varigraph对于植物基因组的变异分析,特别是重复区域或结构变异的分型具有较为优异的表现。
前期研究表明,变异分型软件在超大规模泛基因组图谱上的变异分型能力会明显下降。本研究利用已发表的252个水稻基因组测试了不同大小图谱下的变异分型效果,结果表明Varigraph在大型数据集中的分型效果依然表现出较高的准确性和稳定性。
在异源四倍体油菜、异源六倍体小麦以及同源四倍体马铃薯等基因组上的测试,表明Varigraph同样优于其他软件;Varigraph能在较短时间内处理近1.1亿个变异构成的小麦泛基因组图谱,且结构变异的分型F值超过0.7。Varigraph能实现同源四倍体的变异分型,能较为精确地区分双等位变异下的五种可能的基因型。其中,对于插入和缺失类型的结构变异分型F-值能达到0.73-0.81。
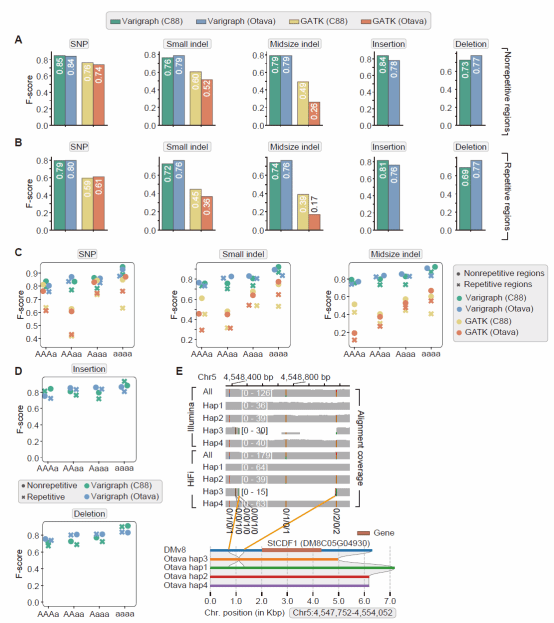
图2 Varigraph和其他基于泛基因组图的分型工具在同源四倍体马铃薯基因组上的分型效果
本研究开发了一种精确且适用范围广泛的基于泛基因组图的变异分型算法Varigraph。Varigraph 是首个能有效利用泛基因组图对同源多倍体进行基因分型的工具,可精确测定等位基因剂量。该研究为植物基因组基因分型提供了一个稳健且准确的解决方案,将推动种质资源精准鉴定、多倍体基因组学等领域的研究。
家庭教师av 已毕业博士研究生杜泽臻为文章第一作者,焦文标教授为通讯作者。课题组研究生贺家宝、肖培轩、博士后胡健兵、家庭教师av 杨宁教授也参与了该研究工作。该工作得到国家自然科学基金面上项目(32270685)、国家自然科学基金优秀青年科学基金(海外)和果蔬园艺作物种质创新与利用全国重点实验室自主培育项目资助。
焦文标教授课题组长期从事植物基因组学研究。课题组以园艺作物为研究对象,基于高通量组学和AI技术,开发生物信息学算法,以解决基因组组装与遗传变异识别、多倍体作物基因组育种体系等领域的关键问题,在Nature Genetics、Molecular Plant、Genome Biology、Nature Communications、Genome Research等期刊发表SCI论文30余篇。
