(图文|王佳诗 编辑|辛西 审核|翟瑞芳)近日,家庭教师av 翟瑞芳副教授团队在Plant Phenomics杂志在线发表题为“PlantCaFo: An efficient few-shot plant disease recognition method based on foundation models”的研究论文,该研究提出一种名为PlantCaFo的高效小样本植物病害识别模型,为植物病害识别领域带来新突破。
精准识别植物病害是筑牢粮食安全防线的关键环节。构建数智化的病害监测预警体系,可显著提升农业防灾减灾能力,是实现“藏粮于技”战略、推动农业全链条减损增效的核心突破点。尽管计算机视觉和人工智能技术在该领域已有长足进步,但现有方法普遍依赖复杂的模型架构和需要海量精确标注的病害图像数据。这种对“大数据”的过度依赖,严重制约了智能识别技术在田间复杂场景下的实际应用潜力。因此,如何利用极其有限的病害样本数据进行高效、准确的识别,成为当前植物保护领域亟待解决的关键难题。
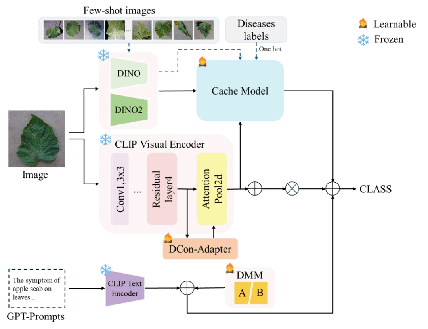
面对农业实践中植物叶片病害数据稀缺的普遍挑战,团队创新性地提出了 PlantCaFo模型。该研究巧妙地以简化的CaFo为基础,创造性利用GPT等生成式人工智能构建了高质量的图像-文本配对数据集,充分融合了 CLIP、DINO 等先进基础模型在视觉与语义理解方面的预训练知识优势,有效弥补了真实病害样本数量的不足,并创新性地设计了 “扩张适配器”(DCon-Adapter) 和 “权重分解模块”(WDM) 。这些设计显著增强了模型捕捉植物病害细微特征的能力,无论是叶片整体的病变模式还是局部的病斑细节,都能被精准提取和分析。
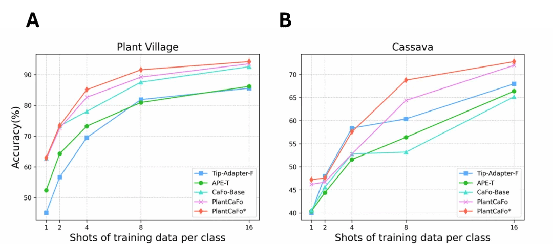
实验表明,在具有简单背景的Plant Village数据集上每类仅使用4幅图像训练就能达到82.63%的识别准确率,比基线高出4.6%;每类使用16幅图像能达到93.53%,比基线高出约1%,显著优于Tip-Adapter-F(85.58%)、APE-T(86.21%)等模型。在复杂自然背景的Cassava数据集上每类使用16幅图像训练的识别效果比基线高出6.8%。
PlantCaFo 的核心价值在于其“小样本、大能力”的特性。 它成功摆脱了传统深度学习方法对大规模标注数据的重度依赖。在仅有少量病害样本的条件下,该模型能充分利用基础模型的预训练知识,快速学习并实现高精度的病害分类。该方法不仅大大降低了技术应用的门槛,还为植物病害识别提供了轻量化技术工具,对作物保护与粮食安全具有重要实践意义。
我校家庭教师av 研究生江雪为论文第一作者,家庭教师av 副教授翟瑞芳为通讯作者,植物科学技术学院杨万能教授参与研究,家庭教师av 硕士研究生王佳诗、谢凯、崔晨曦、杜奥博,博士研究生石祥龙等参与研究工作。该研究得到国家重点研发计划项目的资助。
